現時点で、収束の兆しが見えていないコロナですが、ジョンズ・ホプキンズ大学では、日々の感染者数についての情報を以下のリンク先で公開しています。
https://github.com/CSSEGISandData/COVID-19
この記事では、4/3までの各国の感染者数のデータを元にどのような傾向で増加しているのかをPythonとMatplotlibを使用して可視化してみます。
この記事に掲載されているソースコードには、pandasのデータフレームの加工の方法(行・列のフィルタ、行列の転置、集計など)やMatplotlibを用いたグラフの書き方(アニメーション含む)についての要素が散りばめられているので、よければご参考にしてください。
データの取得から前加工まで
データ読み込み
感染者数データは、上記サイトからコマンドラインでwgetを用いて取得します。(別にcurlとかでも良いです。)
wget https://github.com/CSSEGISandData/COVID-19/raw/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_global.csv
まずはpandasとmatplotlibを使用するためのライブラリをインポートします。
(「%matplotlib inline」はjupyter notebook上でグラフを表示させるための処理です。)
import pandas as pd
%matplotlib inline
import matplotlib.pyplot as plt
取得した感染者数のデータをpandasを使用して読み込みます。
covid_data_org = pd.read_csv("./time_series_covid19_confirmed_global.csv")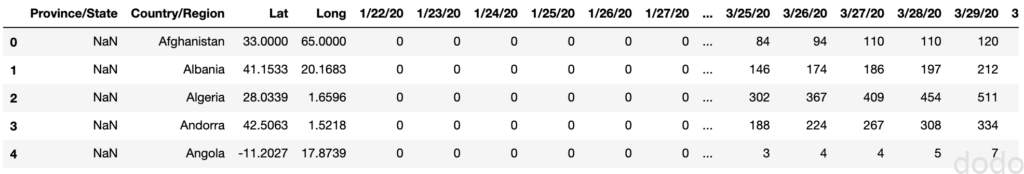 COVID-19感染データ(累計)
COVID-19感染データ(累計)国毎、そして一部の国については州(国の中の地域)毎のレコードとなっており、日付の列にその日までの累計の感染者数が格納されています。
国毎に集計
今回、可視化したいのは国毎のデータなので、同じ国で異なる州は国毎に集計してしまいます。また不要な、州(Province/State)、緯度(Lat)、経度(Long)の列は削除します。
covid_data = covid_data_org.copy()
#州、緯度、経度の列を削除
covid_data.drop(["Province/State","Lat","Long"], axis=1,inplace=True)
#国毎に感染者数を集計
covid_data = covid_data.groupby(['Country/Region'], as_index=False).sum()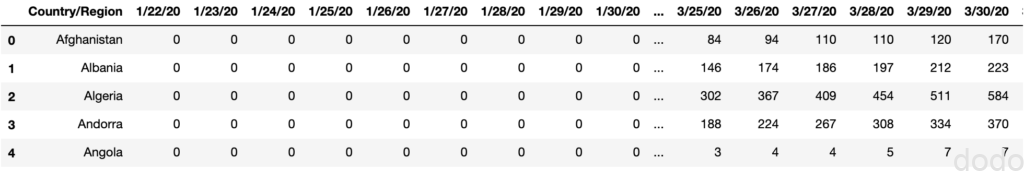 COVID-19感染データ(累計) (国別に加工)
COVID-19感染データ(累計) (国別に加工)可視化する国を絞り込み
国は全部で181国あり、そのままグラフにするとわけわからなくなるので、今回は、感染者数が多いワースト8ヶ国と日本・韓国の合計10ヶ国のみを可視化します。
汎用性を持たせるために、上位何ヶ国か(n)、どの国を含めるか(country_list)を指定してデータを絞り込むための関数を作成します。
def get_covid_data_selected(covid_data, n, country_list):
#TOP n で絞り込み (最後の日付(累計値)の値を基準とする。)
covid_data_selected = covid_data.sort_values(
covid_data.columns[-1], ascending=False)[0:n]
#指定した国を追加
for country in country_list:
covid_data_country = covid_data[covid_data['Country/Region']==country]
covid_data_selected = pd.concat([covid_data_selected, covid_data_country], axis=0)
return covid_data_selected
国と日付を転置
Matplotlibにデータを読み込ませるためには、行が国毎、列が日付毎となっているよりも、行が日付毎、列が国毎になっている方が処理がしやすいです。
なので行と列を転置します。
以下のソースコードでは転置した後、国を列名に設定して、日付をdatetime型のインデックスに変換しています。(日付をdatetime型のインデックスにしておくと、後で1日毎のデータを週毎のデータに集約するような処理で都合が良いのです。)
def get_covid_data_transposed(covid_data):
#転置
covid_data = covid_data.T
#1行目(国名を格納されている)をヘッダーとする
covid_data.columns = covid_data.iloc[0]
#2行目以降(日付毎の観戦数が格納されている)をデータとする。
covid_data = covid_data[1:]
#日付をbaseIndexからdatetimeIndexにする。
covid_data.reset_index(inplace=True)
covid_data.rename(columns={'index': 'date'},inplace=True)
covid_data["date"] = pd.to_datetime(covid_data["date"])
covid_data.set_index('date', inplace=True)
return covid_data
日付毎の感染者数データを生成
取得したデータの数値は、その日付までの感染者の累計数となっています。
日別の感染者数のデータも可視化したいため、日付間のデータの差分を取ることにより、日別の感染者数を持つデータを生成します。
def get_covid_data_diff(covid_data_transformed):
#累計データから差分(日別データ)を算出する。(最初の行は累計データの初期値とする)
covid_data_transformed_diff = covid_data_transformed.diff().fillna(covid_data_transformed.iloc[0])
return covid_data_transformed_diff
解析用のデータを生成
以上、3つの処理(「国を絞り込む」「転置する」「日別のデータを生成する」)をまとめて、累計データと日別データを返す関数を作成します。
def get_covid_data_transformed(covid_data, n, country_list):
covid_data = get_covid_data_selected(covid_data, n, country_list)
covid_data = get_covid_data_transposed(covid_data)
return covid_data,get_covid_data_diff(covid_data)
上記関数を実行して、感染者数ワースト8の国、日本、韓国の感染者数の累計データと日別データを取得します。
# 感染数top 8 + 日本、韓国のデータを解析用に加工して取得
covid_data_total,covid_data_daily = get_covid_data_transformed(
covid_data ,8, ["Japan","Korea, South"])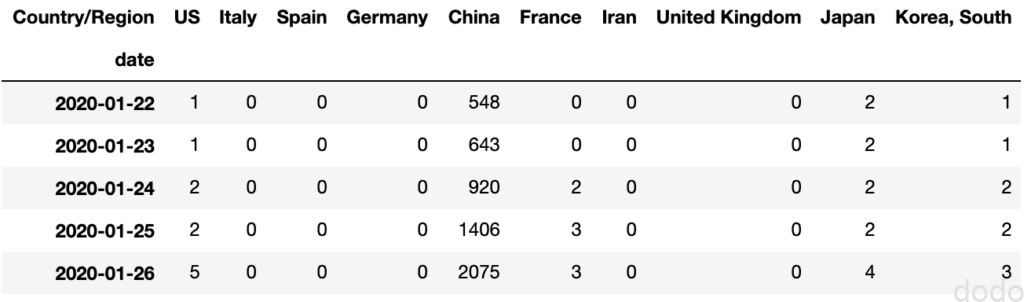 COVID-19感染データ(累計) (国を列、日付を行に変換)
COVID-19感染データ(累計) (国を列、日付を行に変換)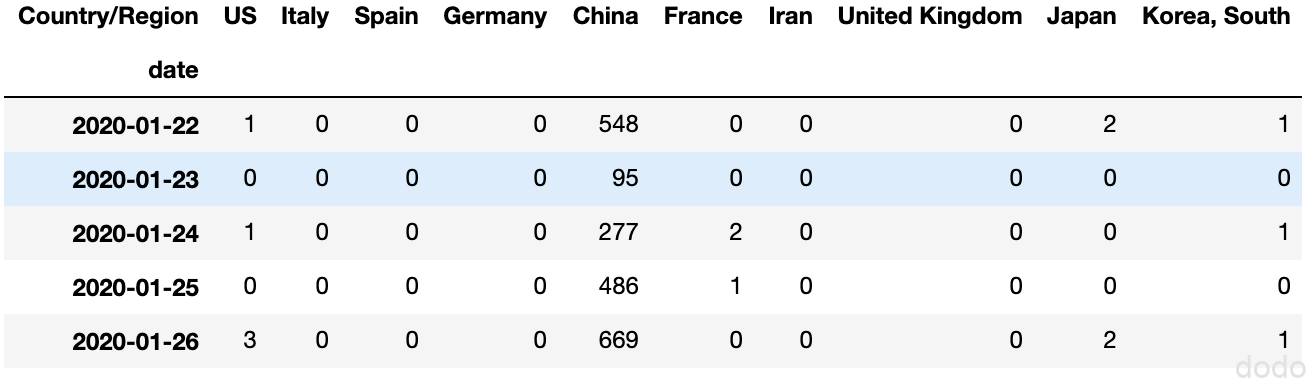 COVID-19感染データ(日別) (国を列、日付を行に変換)
COVID-19感染データ(日別) (国を列、日付を行に変換)可視化(日付時系列)
加工したデータを元に可視化していきます。
前段階として、Matplotlibのデフォルトの文字の大きさを12から15に変更しておきます。(12だとちょっと小さい・・)
plt.rcParams["font.size"] = 15
感染者数(累計)vs 日付
まずは単純に日付と感染者数(累計)でデータをプロットします。
ax = covid_data_total.plot(figsize=(12, 6))
ax.set_title("COVID-19 感染者数(累計)")
plt.savefig("./covid-19-tragectory-1.png", bbox_inches='tight', pad_inches=0.2)
plt.show()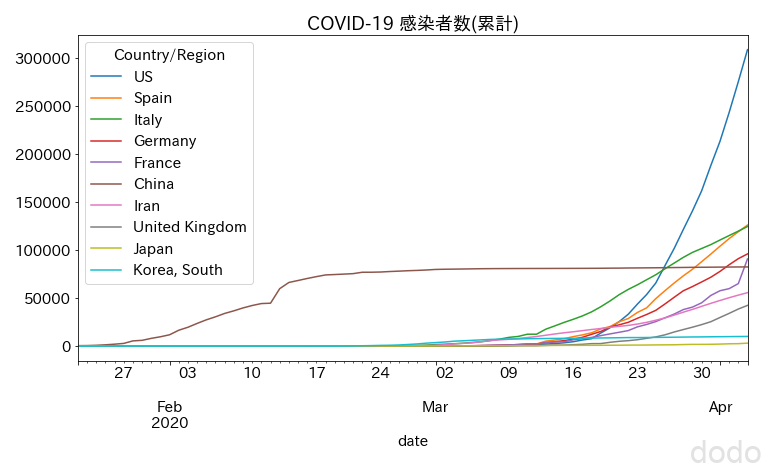 COVID-19感染データ推移(累計)
COVID-19感染データ推移(累計)アメリカ(US)がやばい事は、一目瞭然なのですが、日本はどうか・・となると、アメリカや他の国とスケールが異なりすぎて全然少ないので、大した事ないような気がしてきます。
次に感染者数を対数スケールに変換してプロットしてみましょう。
ax = covid_data_total.plot(figsize=(12, 6))
ax.set_title("COVID-19 感染者数(累計)")
ax.set_yscale("log")
plt.savefig("./covid-19-tragectory-2.png", bbox_inches='tight', pad_inches=0.2)
plt.show() COVID-19感染データ推移(累計) (片対数)
COVID-19感染データ推移(累計) (片対数)感染者数を対数スケールにすると1,10,100,1000・・・と10倍毎に等間隔となるので、スケールが違うデータでも比較しやすくなり、今後の増加傾向が捉えやすくなります。
日本の場合、対数スケールに対して傾きは小さいながらも直線的に上昇しているので、ちょっと油断がならない感じに見えます。
感染者数(日別)vs 日付
次に日別の感染者数をプロットしてみます。
累計の感染者数と同様に通常のスケールと対数スケールの両方で可視化しましょう。
ax = covid_data_daily.plot(figsize=(12, 6))
ax.set_title("COVID-19 感染者数(日別)")
plt.savefig("./covid-19-tragectory-3.png", bbox_inches='tight', pad_inches=0.2)
plt.show()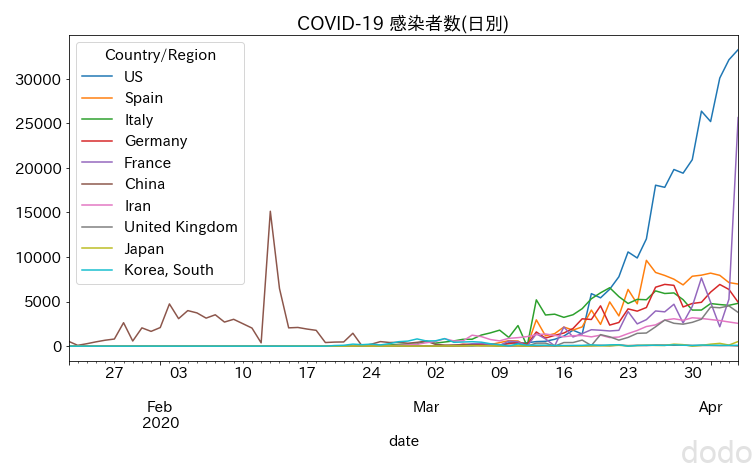 COVID-19感染データ推移(日別)
COVID-19感染データ推移(日別)ax = covid_data_daily.plot(figsize=(12, 6))
ax.set_title("COVID-19 感染者数(日別)")
ax.set_yscale("log")
plt.savefig("./covid-19-tragectory-4.png", bbox_inches='tight', pad_inches=0.2)
plt.show()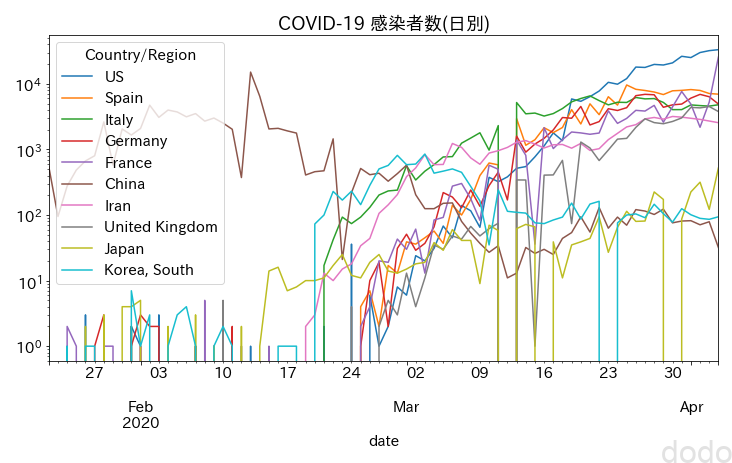 COVID-19感染データ推移(日別) (片対数)
COVID-19感染データ推移(日別) (片対数)対数グラフにした場合、日本も増加傾向にある事はわかりますが、ちょっとわかりづらいかと思います。
日別データが生きてくるのは、次節以降の可視化となります。
可視化(期間vs累計)
次に感染者数(日別)と感染者数(累計)の関係をプロットしたグラフを作成しましょう。
このグラフの利点は時系列が異なる国同士での増加傾向を比較をできる事です。
まずは通常のスケールでプロットします。(縦軸が日別、横軸が累計になります。)
fig = plt.figure(figsize=(12, 6))
ax = fig.add_subplot(1,1,1)
for column_name, item in covid_data_total.iteritems():
ax.plot(item,covid_data_daily[column_name],label=column_name)
ax.set_title("COVID-19 感染者数(日別vs累計)")
#軸ラベル
ax.set_xlabel("感染者数(累計)")
ax.set_ylabel(f"感染者数(日別)")
ax.legend()
plt.savefig("./covid-19-tragectory-5.png", bbox_inches='tight', pad_inches=0.2)
plt.show()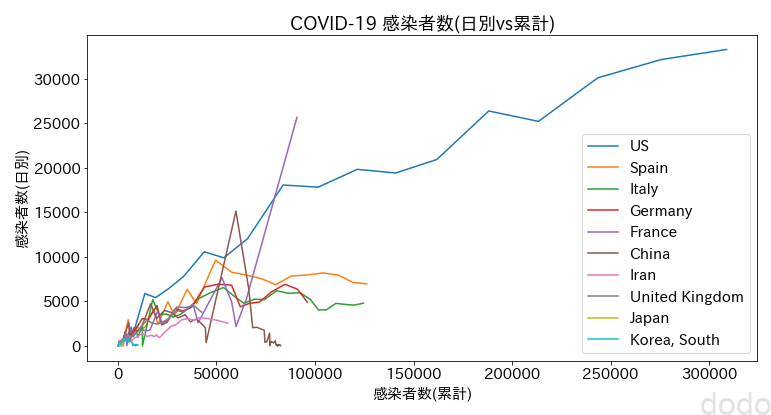 COVID-19感染データ推移(日別vs累計)
COVID-19感染データ推移(日別vs累計)通常のスケールだとやはりアメリカしかよくわからないので、対数スケール(縦横両方)で表示してみます。
fig = plt.figure(figsize=(12, 6))
ax = fig.add_subplot(1,1,1)
for column_name, item in covid_data_total.iteritems():
ax.plot(item,covid_data_daily[column_name],label=column_name)
ax.set_title("COVID-19 感染者数(日別vs累計)")
ax.set_yscale("log")
ax.set_xscale("log")
#軸ラベル
ax.set_xlabel("感染者数(累計)")
ax.set_ylabel(f"感染者数(日別)")
ax.legend()
plt.savefig("./covid-19-tragectory-6.png", bbox_inches='tight', pad_inches=0.2)
plt.show()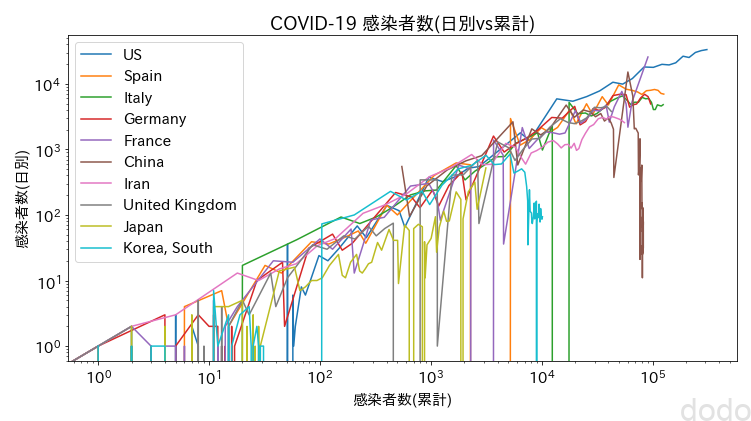 COVID-19感染データ推移(日別vs累計) (両対数)
COVID-19感染データ推移(日別vs累計) (両対数)ばらつきは目立ちますが、どの国も割と同じ傾向で上昇していってるのがわかります。
ばらつきの原因は、日別だとおそらく計測の関係で少ないケース(翌日に繰越してカウントするようなケース)があるからだと思われます。
週別ならばそのばらつきも少なくなると思われるので、週別の感染者数と累計の感染者数をプロットしてみます。
なお汎用性を持たせるために期間を引数として受け取れる関数を作成します。
関数内では引数で渡された期間(period)を元にデータフレームをリサンプルして集計しています。(デフォルトで週(“W”)としています。)
def plot_covid_trajectory(covid_data_total,
covid_data_daily,
period="W",
period_title="週別",
logscale=True):
fig = plt.figure(figsize=(12, 6))
ax = fig.add_subplot(1,1,1)
#累計は最大値でリサンプル
covid_data_total = covid_data_total.resample(period, label='left', closed='left').max()
#期間別は期間内の総和でリサンプル
covid_data_daily = covid_data_daily.resample(period, label='left', closed='left').sum()
#期間別と累計でプロットする。
for count, (column_name, total_data) in enumerate(covid_data_total.iteritems()):
period_data = covid_data_daily[column_name]
ax.plot(total_data,period_data,'o-',label=column_name)
ax.annotate(
column_name, xy = (total_data[-1], period_data[-1]),
size = 15,
color = plt.rcParams['axes.prop_cycle'].by_key()['color'][count])
#タイトル
ax.set_title(f"COVID-19 感染者数({period_title}vs累計)")
#軸ラベル
ax.set_xlabel("感染者数(累計)")
ax.set_ylabel(f"感染者数({period_title})")
#ログスケールかどう
if(logscale):
ax.set_xscale("log")
ax.set_yscale("log")
ax.legend()
plt.savefig("./covid-19-tragectory.png", bbox_inches='tight', pad_inches=0.2)
plt.show()
作成した関数に累計データ、日別データを渡して実行します。
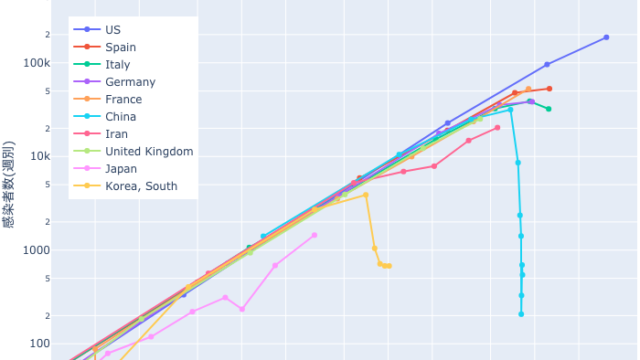
plot_covid_trajectory(covid_data_total,covid_data_daily)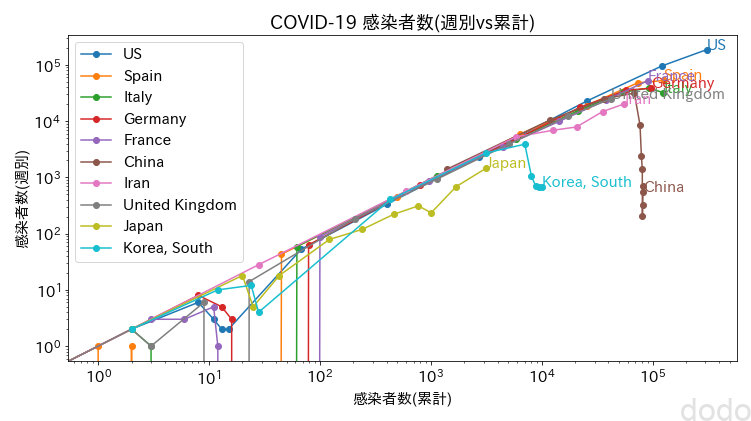 COVID-19感染データ推移(週別vs累計) (両対数)
COVID-19感染データ推移(週別vs累計) (両対数)週別にするとばらつきが少なくなりだいぶ見やすくなったと思います。(なお(●)が週ごとの計測点になります。)
このグラフからわかる事は、感染者数はアメリカ・ヨーロッパが先行こそしているものの、日本もほぼ同じ運命の直線を辿ってる事です・・(ちょっとだけ下にずれてますが・・)
例外として韓国と中国だけがこの直線を脱出してピークアウトしています。(報告されている感染者数が正しいとしてですが・・)
可視化(アニメーション)
最後に、累計・週別の感染者数グラフを時系列(週毎)でプロットしていくようにアニメーション化してみます。
まずは必要なライブラリをインポートします。(なおPillowがインストールされている必要があります。また「%matplotlib notebook」はJupyter notebook上でアニメーションを表示するための処理です。)
%matplotlib notebook
import matplotlib.pyplot as plt
from matplotlib import animation
from matplotlib.animation import PillowWriter
次に、累計・日別の感染者数グラフを作成する関数を改造してアニメーションを表示するための関数を作成します。
詳しい説明は省きますが「plot」という関数を作成して、その中で1週1週のデータを順次プロットしていき、FuncAnimationからコールバックして呼び出して描画させるようにしています。
また描画範囲は50件以上としました。
def plot_covid_trajectory_animation(covid_data_total,
covid_data_daily,
period="W",
period_title="週別",
title_date_format="%Y年%m月%d日週",
logscale=True):
fig = plt.figure(figsize=(12, 6))
#plt.savefigのように保存時にマージンを指定できないので、軸の位置で調整する。
ax = fig.add_axes([0.07,0.1,0.92,0.84]) #left,bottom,width,height
#累計は最大値でリサンプル
covid_data_total = covid_data_total.resample(period, label='left', closed='left').max()
#期間別は期間内の総和でリサンプル
covid_data_daily = covid_data_daily.resample(period, label='left', closed='left').sum()
def plot(data):
# 前のグラフを削除
plt.cla()
#期間別と累計でプロットする。
covid_data_total_tmp = covid_data_total[0:data+1]
covid_data_daily_tmp = covid_data_daily[0:data+1]
for count, (column_name, total_data) in enumerate(covid_data_total_tmp.iteritems()):
period_data = covid_data_daily_tmp[column_name]
ax.plot(total_data,period_data,'o-',label=column_name)
ax.annotate(
column_name, xy = (total_data[-1], period_data[-1]),
size = 15,
color = plt.rcParams['axes.prop_cycle'].by_key()['color'][count])
#タイトル
ax.set_title(
f"COVID-19 感染者数({period_title}vs累計) \
({covid_data_total_tmp.index[-1].strftime(title_date_format)})")
#軸ラベル
ax.set_xlabel("感染者数(累計)")
ax.set_ylabel(f"感染者数({period_title})")
#スケール固定
ax.set_xlim(50, 500000)
ax.set_ylim(50, 500000)
#ログスケールかどうか
if(logscale):
ax.set_xscale("log")
ax.set_yscale("log")
#凡例
ax.legend(loc='upper left')
# 10msごとにplot関数を呼び出してアニメーションを作成
ani = animation.FuncAnimation(fig, plot, interval=500, frames=covid_data_total.shape[0])
ani.save('./covid-19-tragectory.gif', writer='pillow')
plt.show()
上記の関数を実行します。
plot_covid_trajectory_animation(covid_data_total,covid_data_daily)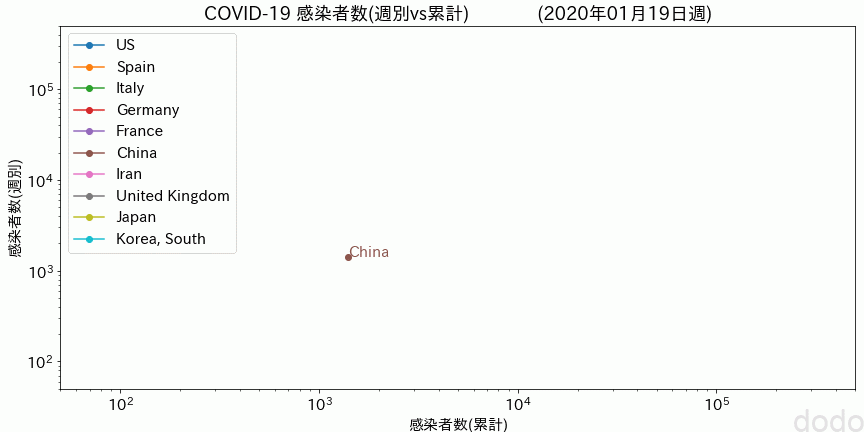 COVID-19感染データ推移(週別vs累計) (両対数/アニメーション)
COVID-19感染データ推移(週別vs累計) (両対数/アニメーション)※Jupyter上でうまくアニメーションが表示されない時は、「plt.close(“all”)」を実行してみてください。他のグラフのplotの影響でうまくいってない可能性があります。
このアニメーションでは1週/0.5秒の速さで描画しています。
よって線の「伸び」が速いほど増加スピードが早い事になり、アメリカはすごい勢いで伸びていってるのがわかります。
なお日本の状況を見ると、基本的に増加傾向にありますが、3月15日週(3月15日〜3月21日)に一時的に感染者数が減っているように見えます。
まとめ
以上、ジョンズ・ホプキンズ大学が提供しているコロナの感染者数データをPython+Matplotlibを使って可視化しました。
技術的な事に関して言えば、以下の要素が上記ソースコードに散りばめられているので、何かのご参考になれば幸いです。
- データフレームのソート
- データフレームの列名による絞り込み
- データフレームの行列転置
- データの集計
- データのカラム化
- データのインデックス化
- datetimeインデックスへの変換
- 時系列データのリサンプリング(日別→週別)
- Matplotlibによる折れ線グラフ作成
- Matplotlibによるアニメーション作成
そしてデータを可視化した内容を見る限り、まだまだ日本も油断ならない感じですね・・
なるべく早く収束する事を祈るばかりです。
なお日別(週別)の感染者数と累計の感染者数でプロットするアイデアについては、以下のサイトを参考にさせて頂きました。(ダッシュボードが非常にわかりやすいのでおすすめです。)
https://aatishb.com/covidtrends/